Des chercheurs norvégiens en médecine légale rapportent avoir eu recours à la généalogie génétique pour faire avancer trois enquêtes criminelles non élucidées. Dans deux affaires, cette technique a permis d’identifier avec succès la personne dont l’ADN avait été retrouvé sur la scène de crime. Selon les auteurs de l’étude, publiée en mars 2025 dans la revue Forensic Science International : Genetics, ces résultats démontrent que cette approche a atteint un degré de maturité suffisant pour identifier une personne inconnue à partir de l’ADN retrouvé sur une scène de crime.
Pour qu’une telle méthode puisse être utilisée dans le cadre d’une enquête policière, il est en effet nécessaire de disposer d’un échantillon biologique contenant une quantité d’ADN exploitable. Encore faut-il que la trace biologique puisse être reliée sans ambiguïté au crime. Cette évaluation doit être réalisée par les autorités policières avec l’appui d’experts en criminalistique.
Bien que la généalogie génétique ait été utilisée pour la première fois au début de l’année 2018, elle s’est fait largement connaître quelques mois plus tard avec l’identification du Tueur du Golden State. Cet ancien policier, Joseph James DeAngelo, a été confondu non pas par son propre ADN, mais par celui de membres de sa famille. C’est un cousin au troisième degré, ayant téléchargé son profil génétique sur le site californien GEDmatch, qui a permis de remonter jusqu’à lui. DeAngelo, auteur de dizaines de meurtres, viols et cambriolages commis en Californie dans les années 1970 et 1980, était resté introuvable pendant quarante-deux ans.
Depuis l’arrestation de Joseph DeAngelo en avril 2018, la généalogie génétique appliquée aux enquêtes criminelles a suscité un vif intérêt, s’imposant comme un outil particulièrement efficace pour résoudre des affaires non élucidées, parfois depuis des décennies. Ce tournant s’explique notamment par le contraste saisissant entre les moyens mobilisés avant l’utilisation de cette méthode et son efficacité une fois mise en œuvre : il avait fallu pendant quatre décennies, 650 enquêteurs, quinze agences de police différentes et près de 10 millions de dollars avant que l’ADN de DeAngelo soit enfin téléchargé sur la base GEDmatch. Une fois cette étape franchie, il n’aura fallu que cinq généalogistes, quelques centaines de dollars et à peine 63 jours pour identifier le suspect.
En Europe, le premier usage documenté de cette méthode remonte à 2019 en Suède pour élucider un double meurtre commis 15 ans plus tôt à Linköping. En 2023, la police australienne l’a utilisée pour résoudre deux cold cases. La même année, les Pays-Bas ont lancé une étude pilote portant sur deux affaires non élucidées. En 2024, la Nouvelle-Zélande y a eu recours dans deux affaires de meurtre remontant à 1980 et 2008.
Aujourd’hui, d’autres pays, dont le Royaume-Uni, la Suède, l’Australie, la Chine, l’Espagne, sont en train d’étudier ou ont déjà mis en œuvre la généalogie génétique dans le cadre d’enquêtes médico-légales (forensic investigative genetic genealogy).
En France, en 2022, la généalogie génétique a permis d’identifier Bruno L., un retraité de 62 ans vivant en Seine-et-Marne, comme l’auteur d’une série de viols commis entre 1998 et 2008 en région parisienne. Malgré la présence répétée de son ADN sur les scènes de crime, son profil n’était pas enregistré dans le Fichier national automatisé des empreintes génétiques (FNAEG). La France a sollicité l’appui du FBI pour mener des analyses via des bases étrangères. Deux parents éloignés ont été retrouvés, permettant de remonter jusqu’à cet individu, qui a reconnu les faits avant de se suicider en prison.
À ce jour, environ cinq cents affaires ont été élucidées grâce à la généalogie génétique utilisée à des fins médico-légales. Cette méthode a permis de résoudre des crimes violents restés longtemps non résolus, d’identifier des restes humains anonymes et même d’innocenter au moins deux personnes.
Actuellement, trois bases de données de généalogie génétique (GEDmatch PRO, FamilyTreeDNA, DNASolves) autorisent explicitement le téléchargement de fichiers de données génétiques issus d’échantillons biologiques à des fins de généalogie génétique appliquée aux enquêtes criminelles.
Rechercher des individus apparentés à l’auteur des faits
La personne à laquelle cet ADN est attribué est désignée comme la « cible ». Dans de nombreux cas, cette cible s’avère être l’auteur des faits, mais plus largement, il peut s’agir de toute personne, vivante ou décédée, susceptible de présenter un intérêt pour l’enquête.
Les trois affaires sur lesquelles Håvard Aanes et ses collègues de l’université d’Oslo ont travaillé concernaient des viols et un meurtre. Dans chaque cas, la police disposait d’un profil ADN exploitable, mais celui-ci n’avait jusqu’alors permis d’identifier aucun suspect.
Dans la première affaire, les enquêteurs recherchent l’auteur de deux viols commis à peu d’intervalle. Un échantillon de sperme recueilli sur l’une des scènes a été utilisé pour les analyses : il contenait de l’ADN à une concentration de 0,4 nanogramme par microlitre, soit 0,4 milliardième de gramme pour un millionième de litre !
La deuxième affaire concerne le viol d’une jeune femme, survenu lors d’un grand rassemblement de fin d’études réunissant plusieurs milliers de lycéens. L’échantillon de sperme analysé dans ce cas contenait de l’ADN à une concentration plus élevée : 8,4 nanogrammes par microlitre.
Le troisième crime remonte à l’été 1999. Le corps d’un linguiste à la retraite est retrouvé deux semaines après sa mort dans son appartement situé en plein centre d’Oslo. Les circonstances du décès, associées au temps écoulé avant la découverte, ont compliqué l’enquête, faisant de ce dossier l’un des rares meurtres non résolus en Norvège. Sur les lieux, la police scientifique a prélevé des cellules épithéliales (provenant de la peau ou des muqueuses). Cet unique échantillon a permis d’extraire de l’ADN à une concentration de 0,2 nanogramme par microlitre.
ADN, profil génétique et criminalistique
Pour comprendre comment la généalogie génétique est utilisée à des fins médico-légales dans le cadre d’enquêtes policières pour identifier un suspect, il faut d’abord savoir que l’idée de base consiste à exploiter l’ADN retrouvé sur une scène de crime, non pas pour remonter directement jusqu’à l’auteur des faits, mais jusqu’à des personnes qui lui sont apparentées.
Avant de voir comment l’ADN peut révéler des liens de parenté, il faut s’intéresser à la manière dont le matériel génétique se transmet de génération en génération.
Notre matériel génétique est composé de 23 paires de chromosomes, soit 46 chromosomes au total. Ces chromosomes sont de longues molécules d’ADN. Si tous les êtres humains partagent plus de 99 % de leur ADN, ce sont les petites différences restantes, appelées variations génétiques, qui nous distinguent les uns des autres et permettent d’établir des liens de parenté.
Sur ces chromosomes, certaines positions précises varient d’un individu à l’autre. Les plus fréquentes et les plus utiles pour l’analyse généalogique sont appelées SNPs (prononcer « snips »), acronyme de Single Nucleotide Polymorphisms, que l’on peut traduire par « polymorphismes nucléotidiques ».
Il s’agit de positions spécifiques de l’ADN où une seule lettre (A, T, C ou G) diffère d’une personne à l’autre. Par exemple, à un emplacement donné, une personne peut avoir un A, une autre un G. Ces points de variation, présents partout dans le génome, servent de petits repères comparables entre individus.
Ces variations (également appelées polymorphismes génétiques) sont extrêmement nombreuses : on en compte plusieurs millions dans le génome humain. Elles sont précieuses pour la généalogie génétique, car elles permettent de comparer les génomes de deux personnes et de repérer des segments identiques, transmis par un ascendant commun.
Mais pour comprendre comment ces segments d’ADN sont transmis à l’identique ou, au contraire, remaniés, au fil des générations, il faut introduire deux notions fondamentales : la méiose et la recombinaison génétique.
Méiose et recombinaison génétique
Lors de la formation des cellules reproductrices (spermatozoïdes et ovocytes) un processus particulier entre en jeu : la méiose.
La méiose est un mécanisme de division cellulaire spécifique qui ne produit pas de simples copies de l’ADN parental, mais génère des combinaisons uniques. Pourquoi ? Parce qu’au cours de ce processus, les chromosomes homologues d’origine paternelle et maternelle s’apparient et échangent entre eux des portions d’ADN. Ce phénomène, appelé recombinaison génétique (ou crossing-over), contribue au brassage génétique.
Cette recombinaison entre chromosomes homologues est essentielle au bon déroulement de la méiose. Elle permet de redistribuer les gènes entre les chromosomes hérités du père et ceux de la mère, produisant ainsi des associations originales. Grâce à ce brassage, chaque spermatozoïde ou chaque ovocyte contient un assemblage unique de matériel génétique. En d’autres termes, les deux chromosomes d’une même paire (l’un paternel, l’autre maternel) peuvent échanger entre eux des fragments d’ADN. Ce processus implique des cassures précises dans les molécules d’ADN, suivies de réparations qui recollent les segments échangés. Ces recombinaisons ont lieu à plusieurs endroits sur chaque paire de chromosomes.
Le résultat ? Chaque cellule reproductrice porte un jeu de chromosomes recombinés, c’est-à-dire un mélange inédit de segments paternels et maternels. Ainsi, deux frères et sœurs reçoivent chacun une combinaison différente de l’ADN de leurs parents : ils partagent en moyenne 50 % de leur matériel génétique, mais pas exactement les mêmes segments.
Ce mécanisme a des conséquences directes sur l’analyse de la parenté génétique. Lorsqu’on compare l’ADN de deux personnes pour déterminer si elles partagent un ancêtre commun, on recherche des segments identiques par descendance : des portions d’ADN transmises sans interruption depuis cet ancêtre commun. Mais plus le nombre de générations entre les deux individus est élevé, plus ces segments initiaux ont été fragmentés par les recombinaisons successives.
Ce processus de recombinaison, essentiel à la diversité génétique, a une conséquence importante pour l’analyse de la parenté : il fragmente progressivement les segments hérités d’un ancêtre. Ainsi, des cousins germains partagent encore de longs segments d’ADN. En revanche, deux personnes dont l’ancêtre commun remonte à quatre ou cinq générations n’auront souvent en commun que de petits fragments, parfois trop courts pour être détectés.
Ainsi, deux frères et sœurs partagent environ 50 % de leur ADN, sous la forme de nombreux segments longs, car ils ont les mêmes parents. Des cousins issus de germains (petits-enfants d’un même couple) partagent, eux, en moyenne 12,5 % de leur ADN. Pour des cousins au cinquième degré, les segments communs sont bien plus petits, plus dispersés, voire absents ou indétectables.
Centimorgans
Les segments d’ADN partagés entre deux individus ne sont pas mesurés en unités physiques mais en centimorgans (cM). Un centimorgan ne correspond pas à une distance réelle sur l’ADN, mais à une probabilité de recombinaison génétique. De manière empirique, 1 cM équivaut en moyenne à environ un million de bases, bien que ce chiffre varie selon la position sur le chromosome, le chromosome concerné, et même le sexe.
Un segment de 1 cM signifie qu’il existe 1 % de probabilité qu’un événement de recombinaison (c’est-à-dire un échange de matériel génétique) survienne à cet endroit précis lors de la méiose, le processus de formation des cellules reproductrices (spermatozoïdes et ovocytes).
En résumé, tout repose sur le fait qu’à chaque génération, l’ADN est brassé par recombinaison lors de la méiose, ce qui fragmente progressivement les segments hérités d’un ancêtre.
En comparant les SNPs, on identifie les segments toujours partagés entre deux personnes, signes d’une origine commune. Leur taille, exprimée en centimorgans, permet de remonter dans le temps jusqu’à cet ancêtre. Plus les segments communs sont longs et nombreux (mesurés en centimorgans), plus le lien de parenté est proche.
C’est ainsi que l’on peut, grâce à quelques millilitres de salive et à l’analyse de quelques centaines de milliers de SNPs, reconstituer des liens familiaux.
Faire parler l’ADN pour reconstruire des liens familiaux
En somme, c’est la compréhension fine de la méiose et de la recombinaison qui permet aujourd’hui aux chercheurs en généalogie génétique de remonter le fil des parentés.
En analysant la taille et la répartition des segments d’ADN partagés, il devient possible de reconstituer des liens familiaux, y compris plusieurs décennies après les faits.
Une fois compris le mécanisme de transmission et de fragmentation des segments d’ADN au fil des générations, il devient possible d’exploiter ces informations pour identifier un individu inconnu à partir de son profil génétique. Celui-ci est défini par l’analyse d’un grand nombre de SNPs.
Comparer le profil SNP de la cible à ceux conservés dans des bases généalogiques
Mais comment remonter jusqu’à un individu à partir d’un simple échantillon biologique (sang, salive, sperme, cellules) retrouvé sur une scène de crime, si son profil génétique ne figure pas dans la base de données de la police scientifique ?
La première étape consiste à rechercher, dans d’autres bases de données, des profils génétiques présentant un lien de parenté avec l’ADN inconnu. En effet, ces bases existent bel et bien : elles sont constituées à partir des résultats de tests réalisés, à titre personnel, par des millions d’individus curieux d’en savoir plus sur leur ascendance, leur état de santé ou leurs origines.
Pour cela, ces personnes reçoivent un kit, souvent acheté en ligne, qui leur permet de prélever leur ADN, par exemple en frottant l’intérieur de la joue avec un grand coton-tige. Le prélèvement est ensuite envoyé à une entreprise spécialisée située aux Etats-Unis qui analyse les données génétiques. En retour, elle fournit des informations sur l’origine géographique et ethnique de la personne, et peut suggérer d’éventuels liens de parenté avec d’autres utilisateurs ayant réalisé le même test.
Ce sont précisément ces bases de données génétiques qui peuvent, sous certaines conditions strictes, être exploitées par les forces de l’ordre – comme le FBI aux États-Unis – pour savoir s’il existe dans ces fichiers un profil SNPs apparenté, de près ou de loin, à celui de l’individu recherché par la police, la « cible ».
En France, les tests ADN à visée généalogique sont interdits. L’article 226-28-1 du Code pénal prévoit une amende de 3 750 euros pour toute personne qui y recourt à des fins personnelles ou dites « récréatives ». Il est donc illégal de faire analyser son ADN pour retracer ses origines familiales ou compléter son arbre généalogique, mais dans les faits, cette interdiction est largement contournée. On estime que 1,5 à 2 millions de Français ont déjà effectué un tel test. À ce jour, aucune amende n’a été infligée à un particulier pour ce motif. La méthode est simple : commander un kit sur un site étranger, le faire livrer dans un autre pays européen, puis se le faire réexpédier en France.
Correspondances génétiques
Lorsqu’un ou plusieurs segments d’ADN identiques par descendance sont détectés entre le profil inconnu de la « cible » et un individu enregistré dans la base de données génétiques, cela signifie qu’ils partagent un ancêtre commun, plus ou moins lointain. Ces correspondances sont appelées matches ou hits en anglais — autrement dit, des profils génétiques compatibles.
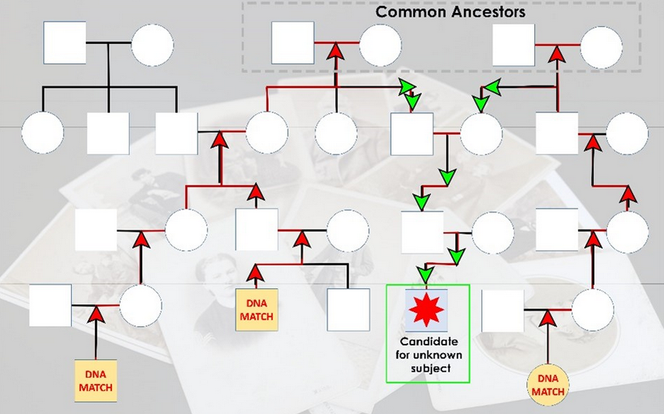
Phase ascendante : remonter vers les ancêtres
Dans un premier temps, on remonte la généalogie des « hits », ces individus dont l’ADN partage une portion significative avec celui de la « cible ». L’objectif est d’identifier les ancêtres communs entre la cible et ces proches génétiques.
Lorsqu’un même segment d’ADN est partagé par plusieurs personnes, en l’occurrence la « cible » et d’autres profils, on parle de triangulation, signe probable d’un héritage commun provenant d’un ancêtre partagé.
Pour chaque correspondance génétique, l’étape suivante consiste à déterminer l’ancêtre commun le plus récent, appelé MRCA (Most Recent Common Ancestor). Il s’agit de la personne, ou du couple, à partir de laquelle la cible et la correspondance génétique descendent.
Phase descendante : explorer les descendants
Une fois cet ancêtre commun identifié, on change de direction et on redescend dans la généalogie en partant de lui : on reconstitue son arbre généalogique descendant jusqu’à aujourd’hui. L’objectif est cette fois d’identifier tous ses descendants, c’est-à-dire les individus susceptibles d’être la cible recherchée.
Cependant, cette correspondance seule ne suffit pas à identifier précisément l’individu recherché : un MRCA peut en effet avoir des dizaines, voire des centaines de descendants potentiels.
Pour affiner la recherche, on croise plusieurs correspondances génétiques issues de personnes partageant un segment d’ADN avec l’individu inconnu, même si elles ne descendent pas forcément du même ascendant. Ce croisement d’informations s’appelle en anglais une intersection. On porte une attention particulière à ces intersections : il s’agit de points où deux branches généalogiques différentes, reliées à des ancêtres distincts, convergent en un même couple ou groupe de descendants. Ces croisements permettent de resserrer la zone de recherche. C’est donc dans ce groupe que la « cible » est logiquement susceptible de se trouver.
Des critères pour réduire la liste des suspects
À ce stade, l’enquête quitte le domaine purement génétique pour s’appuyer sur des méthodes plus classiques. Il s’agit d’examiner les candidats afin de déterminer lequel correspond le mieux au profil recherché.
Plusieurs critères permettent d’éliminer des personnes incompatibles. L’origine géographique ou ethnique estimée de l’ADN – par exemple, européen du Nord, Afrique de l’Ouest ou Asie de l’Est – sert à exclure ceux qui ne correspondent pas à ce profil. On prend aussi en compte l’âge de la cible. En effet, certains marqueurs se modifient chimiquement au fil de la vie, ce qui permet d’estimer l’âge biologique de la personne. Ainsi, un ADN masculin jeune éliminera automatiquement les candidats trop âgés ou trop jeunes.
Enfin, le sexe et d’autres caractéristiques physiques estimées à partir de l’ADN, comme la couleur des yeux, des cheveux ou la pigmentation de la peau, peuvent aussi guider cette sélection.
Confirmation par un test ADN classique
Grâce aux différents critères de filtrage, la liste des suspects est réduite à un petit nombre de personnes, voire à un seul individu. L’étape finale consiste alors à confirmer formellement l’identité en comparant directement l’ADN du suspect à celui retrouvé sur la scène de crime.
Cette vérification repose sur une méthode classique d’analyse génétique, le profilage STR (Short Tandem Repeat), qui permet d’établir une correspondance précise entre les profils génétiques. Si la personne est décédée, il est possible de confirmer son identité en testant ses proches.
Revenons aux trois affaires criminelles dans lesquelles les enquêteurs ont eu recours à la généalogie génétique. Une fois les SNPs identifiés, les profils génétiques ont été téléchargés sur les serveurs de GEDmatch Pro et FamilyTreeDNA (FTDNA), afin de rechercher d’éventuels profils apparentés. GEDmatch Pro compte environ 600 000 profils, tandis que FTDNA en revendique plus de 1,4 million — mais les Norvégiens n’y sont que faiblement représentés, ce qui limite la portée des recherches dans ce contexte.
Les recherches ont cependant généré un trop grand nombre de correspondances (ou hits), rendant l’analyse difficile. Pour y remédier, les chercheurs ont développé un programme informatique permettant de filtrer les résultats, en écartant les correspondances trop fréquentes ou peu informatives, et en ne conservant que les plus pertinentes. Une fois le profil validé par GEDmatch, l’identification des correspondances a pris moins de 24 heures.
Les chercheurs ont ensuite étudié les segments d’ADN partagés afin de repérer ceux communs à plusieurs personnes. Ces segments, appelés « triangulés », indiquent qu’un ancêtre commun est probablement à l’origine de cet ADN, ce qui aide à remonter la piste familiale.
Grâce au kit ForenSeq DNA Signature Prep Kit (Verogen), composé de nombreux marqueurs génétiques, les chercheurs ont pu prédire le phénotype du suspect, à savoir certaines de ses caractéristiques physiques, comme la couleur des yeux, des cheveux, ainsi que son ascendance biogéographique, c’est-à-dire la région du monde d’où proviennent ses ancêtres. L’ensemble de ces marqueurs a été analysé par séquençage, puis grâce à un logiciel dédié.
Ces informations ont permis d’écarter certains individus dont les caractéristiques ne correspondaient pas au profil génétique prédit. Dans un des cas, un trait physique rare s’est révélé particulièrement déterminant. Ainsi, dans la première affaire, les analyses suggéraient une personne aux yeux marron, aux cheveux bruns et d’origine européenne. Dans la deuxième, la cible devait avoir les yeux marron, les cheveux blonds et également une ascendance européenne. Enfin, dans la troisième affaire, le profil correspondait à une personne aux yeux bleus, aux cheveux roux et d’origine européenne. Dans les deux cas où l’individu recherché a pu être identifié, les prédictions se sont avérées justes.
Les chercheurs ont également eu recours à des analyses dites de biogéographie génétique, qui permettent d’estimer les origines géographiques d’un individu à partir de son ADN. Les sites commerciaux FTDNA et GEDmatch PRO proposent aussi ce type d’analyse.
Dans les deux affaires résolues, la personne recherchée avait des ancêtres récents, sur les trois à cinq dernières générations, nés hors de Norvège. Ces origines non norvégiennes laissaient des traces génétiques suffisamment nettes pour orienter efficacement l’enquête généalogique.
Par exemple, si l’un des candidats avait un ancêtre italien, mais que l’ADN de la cible ne montrait aucune trace d’ascendance italienne, cette piste était jugée peu probable. À l’inverse, si le profil génétique indiquait une composante « Europe centrale » et que l’arbre généalogique d’un candidat révélait un ancêtre originaire de cette région, cette branche devenait prioritaire. La même logique a été appliquée à l’échelle nationale : un ADN pointant vers le sud de la Norvège permettait d’écarter les lignées exclusivement issues du nord. Il convient toutefois de rester prudent, estiment les auteurs. En effet, les méthodes d’analyse biogéographique utilisées par FTDNA et GEDmatch PRO n’ont pas encore été validées par des publications scientifiques évaluées par des pairs.
Pour reconstruire les arbres généalogiques à partir des correspondances ADN identifiées, les chercheurs se sont appuyés sur plusieurs sources : le registre national de la population, les registres d’église, les recensements, ainsi que diverses ressources numérisées mises à disposition par la Bibliothèque nationale. Ils ont également consulté des bases de données généalogiques comme Geni et MyHeritage, mais uniquement pour exploiter les informations généalogiques publiques disponibles — sans recourir à leurs outils d’analyse ADN.
Pour identifier les correspondances ADN et retracer des liens familiaux, les chercheurs ont utilisé le Registre national de la population norvégien, contenant des données sur les naissances, décès, mariages, adresses, etc. Ce registre est partiellement numérisé depuis 1964 et entièrement depuis les années 1990, avec une qualité de données décroissante pour les périodes plus anciennes.
Les registres paroissiaux, utilisés jusqu’en 1942, remontent à 1623 et ont aussi servi de registre de population. Pour combler les lacunes entre les différentes sources, les chercheurs ont eu recours aux recensements historiques (notamment ceux de 1920, 1910, 1900, 1891, 1875, 1865 et 1801), ainsi qu’à des archives non publiques, accessibles par l’intermédiaire d’archivistes.
La Bibliothèque nationale a fourni un appui précieux grâce à la numérisation de l’intégralité des collections de journaux locaux (avis de décès, portraits biographiques), ainsi que de nombreux ouvrages d’histoire locale et de généalogie. Des bases de données généalogiques ouvertes comme Geni et MyHeritage ont été utilisées pour obtenir des indices mais leur fiabilité limitée et leur accès payant ont restreint leur usage.
Grâce à la richesse des archives norvégiennes (état civil, recensements, registres paroissiaux), les chercheurs ont pu identifier la plupart des personnes vivant en Norvège partageant un lien génétique avec l’ADN analysé et reconstituer leur généalogie sur 4 à 6 générations. En revanche, l’identification des correspondances vivant à l’étranger, notamment aux États-Unis, s’est révélée plus complexe. L’émigration massive de Norvégiens vers les États-Unis à la fin du XIXe et au début du XXe siècle fait qu’un Norvégien d’aujourd’hui peut avoir des centaines de cousins éloignés outre-Atlantique.
Dans deux affaires, l’âge de la personne recherchée était inconnu. Dans une autre, il se situait dans une fourchette étroite, ce qui a largement facilité l’identification. Cela montre l’intérêt de développer des méthodes fiables d’estimation de l’âge à partir de l’ADN.
Fait notable : dans les deux affaires élucidées, les suspects vivaient à moins de 10 km du lieu du crime au moment des faits.
Dans la première affaire, les premières correspondances génétiques obtenues via GEDmatch Pro et FTDNA étaient trop éloignées et peu fiables pour lancer une enquête généalogique : la plupart concernaient des personnes non norvégiennes, partageant de très courts segments d’ADN, sans lien clair. Le profil génétique complexe et multiethnique de la personne recherchée compliquait encore l’analyse. En revanche, une estimation de son origine géographique ancestrale a pu être établie. Ces indices ont permis d’orienter l’enquête, qui est toujours en cours.
Dans la deuxième affaire, les correspondances génétiques initiales partageaient moins de 50 centimorgans, témoins des liens parentaux très éloignés, parfois liés à un petit village norvégien, avec quelques connexions aux États-Unis.
Grâce à GEDmatch Pro, une correspondance plus proche a été trouvée, partageant 236 cM avec la personne recherchée. Les chercheurs n’ont mis que 10 à 15 heures à identifier le candidat après réception des résultats de GEDmatch Pro. L’arbre généalogique construit comptait alors 319 membres, dont 185 vivants.
Cette personne avait plusieurs demi-cousins en Norvège, certains vivant près du lieu du crime. L’un d’eux présentait un profil génétique et un phénotype cohérents, incluant une ascendance partielle d’Europe centrale.
Un test de profilage STR a confirmé que cette personne était bien la source de l’ADN retrouvé. L’affaire est toujours en cours. Le procès n’a pas encore eu lieu.
Dans la troisième affaire, les recherches ont commencé avec FTDNA, trouvant plusieurs correspondances génétiques entre 50 et 102 cM, mais une avancée majeure est survenue quatre mois plus tard grâce à une correspondance de 87 cM sur GEDmatch Pro, liée à un Américain d’ascendance norvégienne. Toutes les correspondances avaient des ancêtres issus d’un même village norvégien. Les enquêteurs ont alors émis l’hypothèse qu’il existait trois à quatre générations d’écart avec la personne recherchée, ce qui a permis d’orienter l’enquête vers plusieurs branches familiales.
Des tests ADN ciblés ont été réalisés. Plusieurs personnes ont ainsi volontairement donné un échantillon ADN de référence, une seule ayant refusé. Les résultats obtenus ont permis d’écarter certaines branches familiales, tout en recentrant les recherches sur une branche de demi-cousins.
Il ressort de l’enquête que l’individu recherché est décédé. Le coupable ne pourra donc jamais être jugé pour les faits commis. Il s’avère qu’il était connu des services de police et que son ADN figurait dans leurs archives. Celui-ci a été soumis à un profilage STR, une méthode qui consiste à analyser des régions spécifiques du génome, variables d’un individu à l’autre, afin d’établir d’éventuels liens de parenté. Le profil génétique obtenu correspondait à celui de l’ADN retrouvé sur la scène de crime.
Au moment de la résolution de l’affaire, les enquêteurs avaient consacré entre 2 000 et 3 000 heures de travail sur une année et l’arbre généalogique compilait 3 785 individus, dont 1 656 vivants.
L’expérience norvégienne n’est pas une exception. Tout récemment, c’est au Canada que la généalogie génétique a permis de résoudre un cold case vieux de dix-sept ans. Le 17 septembre 2025, le Service de police de la Ville de Montréal (SPVM) a annoncé avoir élucidé, grâce à cette tenchique, le meurtre d’une jeune femme de 26 ans, assassinée en 2008 dans son appartement. Les analyses ADN ont conduit à l’identification de l’auteur : un homme décédé en 2021 dans un centre de détention, où il purgeait une peine pour vols qualifiés et tentatives de meurtre. Aucun lien familial ne le reliait à la victime. Il aurait pris contact avec elle quelques jours avant le drame, après qu’elle eut mis sa voiture en vente sur un site de petites annonces en ligne.
Enjeux éthiques, légaux, sociétaux
Ces dernières années, de nombreux travaux se sont intéressés à l’usage des données génétiques dans le cadre de la généalogie génétique à des fins d’enquête. Ils ont exploré plusieurs questions sensibles : progrès techniques, enjeux sociétaux (notamment le respect de la vie privée), préoccupations du public – qu’il s’agisse du consentement, de l’information ou de la participation volontaire à des tests ADN ciblés -, sans oublier les défis juridiques que cette méthode pose aux forces de l’ordre.
Pour obtenir le soutien du public et prévenir tout usage abusif de la généalogie génétique, il est essentiel de suivre des lignes directrices strictes définissant quand et comment recourir à cette méthode, estiment les chercheurs norvégiens. « Comme pour tout travail utilisant des données sensibles, agir avec professionnalisme et intégrité envers les personnes concernées est indispensable. Le respect rigoureux de ces principes, associé aux progrès scientifiques continus, sera déterminant pour la mise en œuvre et l’acceptation de cette approche dans les enquêtes criminelles les plus graves », concluent-ils.
Pour en savoir plus :
Aanes H, Vigeland MD, Star B, et al. Heating up three cold cases in Norway using investigative genetic genealogy. Forensic Sci Int Genet. 2025 Mar ;76 :103217. doi : 10.1016/j.fsigen.2024.103217
Wang M, Chen H, Luo L, et al. Forensic investigative genetic genealogy : expanding pedigree tracing and genetic inquiry in the genomic era. J Genet Genomics. 2025 Apr ;52(4) :460-472. doi : 10.1016/j.jgg.2024.06.016
Guerrini CJ, Robinson JO, Elsaid MI, et al. FIGG at 5 : An update on U.S. public perspectives on forensic investigative genetic genealogy five years after its introduction to criminal investigations. Forensic Sci Int. 2025 Feb ;367 :112372. doi : 10.1016/j.forsciint.2025.112372
Tuazon OM, Wickenheiser RA, Ansell R, et al. Law enforcement use of genetic genealogy databases in criminal investigations : Nomenclature, definition and scope. Forensic Sci Int Synerg. 2024 Feb 8 ;8 :100460. doi : 10.1016/j.fsisyn.2024.100460
Gurney D, Press M, Moore C, et al. The need for standards and certification for investigative genetic genealogy, and a notice of action. Forensic Sci Int. 2022 Dec ;341 :111495. doi : 10.1016/j.forsciint.2022.111495
Glynn CL. Bridging Disciplines to Form a New One : The Emergence of Forensic Genetic Genealogy. Genes (Basel). 2022 Aug 1 ;13(8) :1381. doi : 10.3390/genes13081381
Dowdeswell TL. Forensic genetic genealogy : A profile of cases solved. Forensic Sci Int Genet. 2022 May ;58 :102679. doi : 10.1016/j.fsigen.2022.102679
Tillmar A, Fagerholm SA, Staaf J, et al. Getting the conclusive lead with investigative genetic genealogy – A successful case study of a 16 year old double murder in Sweden. Forensic Sci Int Genet. 2021 Jul ;53 :102525. doi : 10.1016/j.fsigen.2021.102525
Wickenheiser RA. Forensic genealogy, bioethics and the Golden State Killer case. Forensic Sci Int Synerg. 2019 Jul 12 ;1 :114-125. doi : 10.1016/j.fsisyn.2019.07.003

